引言
顾名思义,分子诊断主要是通过多种方法来评估相关分子、进而分析送检标本中的遗传学改变。作为分子诊断的执行者,病理医师应了解分子检测方法并掌握其优缺点,以更有针对性的将其用于实际工作。根据《Surg Pathol Clin》杂志2021年刊发的分子病理系列文章,我们将为大家编译介绍最常用的分子检测方法,本期请看DNA基础上的检测方案。
【关键词】:分子诊断,预分析方法,二代测序(NGS),聚合酶链反应
【提要】
分析前注意事项,包括样本采集、储存和运输,以及样本评估,对于确保分子诊断测试的成功至关重要
建议对小样本进行组织分类和最大化分子分析材料
分子方法,例如基于测序、基于杂交或聚合酶链
基于反应的具有不同的敏感性和特异性,适当的使用取决于应用
设计分子检测菜单需要考虑生物标志物、患病率、样本类型、和测试方法的局限性。
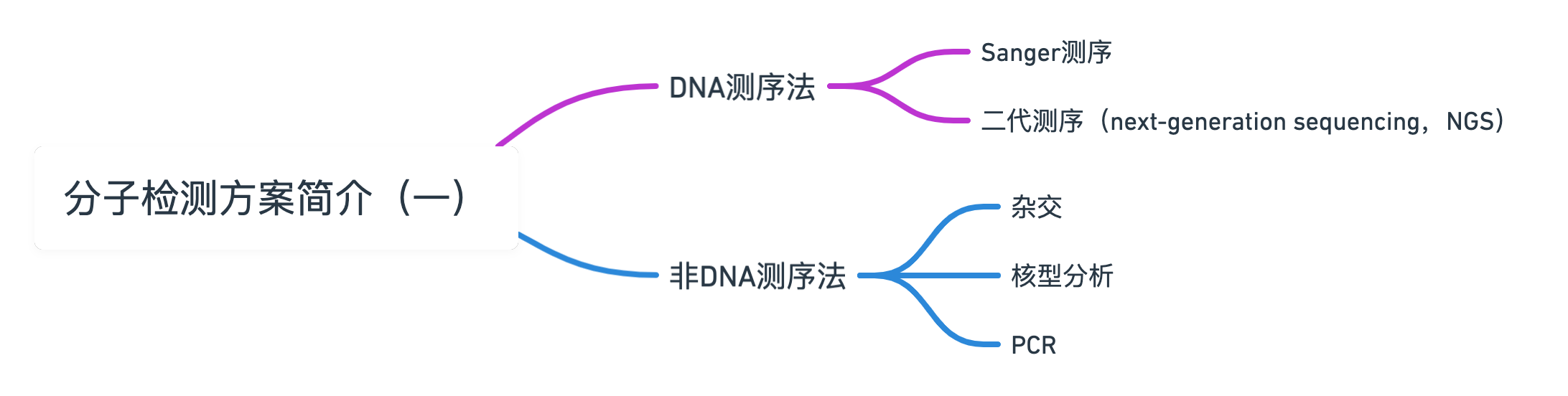
分子检测方法涉及的分子包括DNA、RNA、蛋白,相关方法可检测序列改变、结构变化、拷贝数改变、核酸的共价修饰及蛋白的改变。具体可根据检测的分子类型、或被检测改变的类型进行分类。就DNA基础上的检测来说,具体可分为测序法及非测序的相关方法。
DNA测序法
有多种方法可检测DNA分子,具体可分为测序为主的方案和非测序为主的方案,前者评估靶向DNA分子的全部核苷酸序列。临床常用的测序检测主要有两种,分别是Sanger测序和二代测序(next-generation sequencing,NGS)。
Sanger测序也称为双脱氧测序,是一种有效获取DNA序列信息的方法。该方法是在有脱氧核苷酸(deoxynucleoside triphosphates,dNTPs)分子和双脱氧核苷酸(dideoxynucleoside triphosphates,ddNTP)的情况下扩增DNA片段。ddNTP整合入延长的DNA分子后,延长反应终止。终止点沿着DNA分子全长随机出现且有重复,因此会有很多终止点相同的片段。然后将这些碎片按照大小分开,ddNTP含有对应于结合碱基(A、T、C或G)的荧光标记,可以通过仪器检出并与片段长度相结合。通过这种方法,就可以“读”出特异性序列。
Sanger测序的优点在于耗时短,可以“读”的序列长度要比NGS更长,且可以在检测区域检出单个核苷酸的变化、插入或缺失。不过,Sanger测序的敏感性有限,一般需样本中至少有10%-20%的待测序列才可以。由于癌中很多序列具有异质性,因此需标本中的肿瘤细胞比例至少20%-40%才可以通过该方法准确检出相关改变。此外,Sanger测序的分子是大量“阅读”所得(即所有大小相同的片段同时解读),因此可能很难确定、或无法确定同一Sanger测序的同一DNA条带中、或不同DNA条带中存在多种变异。
最近几年,临床肿瘤实验室已广泛应用NGS,该方法又称为大规模平行测序(massively parallel sequencing)。尽管不同的NGS平台所用方法及试剂有所不同,但其基本原则是相似的。NGS的第一步称为文库构建(library generation)和靶向富集(target enrichment)。
NGS的优势在于敏感性极高,如果采用唯一分子标识符(unique molecular identifiers,UMIs)的方法,可检出1%以下的目标DNA序列。此外,如果相关变异在同一被测分子中极为接近,这一方法可以评估相关突变是在同一DNA条带、还是在不同DNA条带。NGS还可同时分析多个靶向区域,而Sanger测序仅可检测单一区域。不过,NGS相比Sanger测序来说,需要较多的DNA、所需时间较长、单次检测费用更高。需要注意的是,尽管单次Sanger测序反应更快、费用更低,但对于每个碱基的费用来说,取决于靶向区域的大小,因此NGS可能性价比更高。
非DNA测序法
针对DNA的遗传学检测还有非DNA测序的方法,如杂交、核型分析、PCR基础上的方法等。
杂交法的检测中,荧光标记的短DNA序列(即所谓的探针)与特异性靶点互补,可与特异性DNA靶点结合或杂交。这一荧光原位杂交检测方法中,这类探针有多种应用:可以通过和对照信号中(如ERBB2或CNE17)荧光信号的数量进行比较来计算拷贝数改变;可以在靶点区域两端标记不同颜色来证实某一基因的断裂(如MYC断裂),或在配体基因标记不同颜色、通过寻找重叠信号来确定融合基因的融合配体(如BCR-ABL1融合)。荧光原位杂交对于所靶向的特异性改变来说,快速且高敏,但部分变异的改变可能会漏诊(尤其小于荧光原位杂交探针大小的情况下、或某些特殊改变),且每一次检测仅可检测极少数靶点。
染色体微阵列(chromosomal microarray,CMA)是通过每一种可能等位基因的探针、在基因组内千千万万个单核苷酸多态性(single nucleotide polymorphisms,SNPs)中进行检测。每一个位置的每一种探针荧光信号确定SNP的基因型,并确定患者的每一个位置是纯合型、还是杂合型。通过对整个基因组的观察,还可以在极高分辨率水平上(15kb)得出拷贝数改变的信息,且可以获得单个位点的基因型特异信息。CMA在极高分辨率水平上对全基因组进行评估,但由于所用是短探针,一般无法检出平衡易位,因为大量DNA存在时平衡易位并无基因组净值变化。
核型分析是评估基因组改变的最成熟方案,且在一次检测中观察全部基因组。该方法是将活细胞进行培养,使其同步化并停止在细胞周期的分裂间期。然后在玻片表面将细胞轻轻的裂解并染色(一般是Giemsa染色),以获取染色体的条带。每一条染色体都有独特的条带分布,有经验的技师及遗传学专家可以通过解读这些条带分布来描述整个基因组的结构改变、拷贝数改。这一方法耗时3-7天,具体取决于培养细胞的生长速度;且该方法的分辨率低,一般可见的染色体条带对应10Mb大小DNA。
PCR基础上的检测方法有多种,是通过PCR反应的特异性来检测相关改变。最简单的是有突变序列存在时的等位基因特异性扩增方法获取PCR产物,无靶向突变等位基因则无法扩增。还可以通过设计引物来扩增特异性易位,但这一做法更多见于RNA模板检测。PCR的一种定量方法是数字PCR(digital PCR,dPCR),其模板DNA稀释并分割为无模板分子(0)或一个模板分子(1),因此而得名“数字”。临床实验室更常见的是数字液滴PCR(digital droplet PCR,ddPCR),该方法中,模板DNA稀释并乳化至不含(即0)或含一个(即1)模板DNA分子的液滴。然后针对特异基因型进行PCR反应。定量化的报告,可以是每单位容积内特殊基因型液滴的数量,也可以是含DNA液滴的总数(及变异型与正常之和)。PCR检测方法快速、且价格相对低廉,同时对于检测的变异来说敏感性高:实时荧光定量PCR及ddPCR可检出万分之一至十万分之一的变异,常规用于微小或可测量病变的检测及ctDNA评估。尽管有上述优点,但PCR的方法仅可用于评估已知变异,且多路检测的能力有限,因此一次反应仅能针对4-8个靶向变异。
此前所述的DNA检测方案是针对特异性DNA序列、结构或拷贝数;现在甲基化的检测方法可检出胞嘧啶核苷酸中甲基的表观共价结合。这些方法也是用可以裂解或不可裂解甲基的特异性限制酶、或者用亚硫酸氢盐反应来转化未甲基化的尿嘧啶碱基,然后再进行序列特异性检测(如PCR或测序)而确定特异性碱基的甲基化情况。
未完待续
参考文献
Ewalt MD, Hsiao SJ. Molecular Methods: Clinical Utilization and Designing a Test Menu. Surg Pathol Clin. 2021;14(3):359-368.
doi:10.1016/j.path.2021.05.001